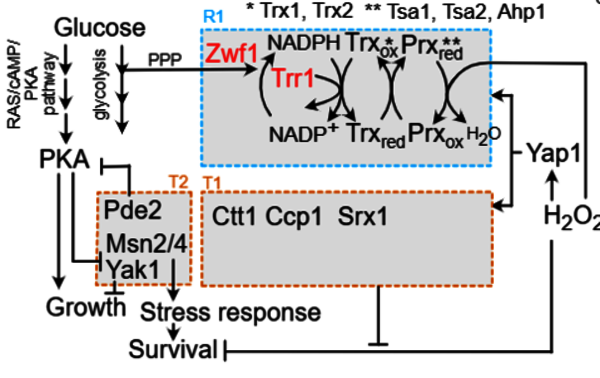
A Trade-Off between Stress Resistance and Tolerance Underlies the Adaptive Response to Hydrogen Peroxide
Basile Jacquel, Bor Kavčič, Audrey Matifas, Thomas Julou, Gilles Charvin Cell Systems 16, 101320 (2025) - DOI: 10.1016/j.cels.2025.101320 Dans ce travail, en collaboration avec les laboratoires de Thomas Julou et Bruce Morgan, nous avons étudié l'interaction mécanistique entre les stratégies…
