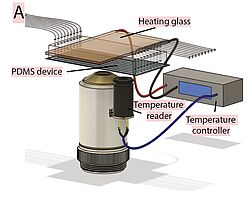
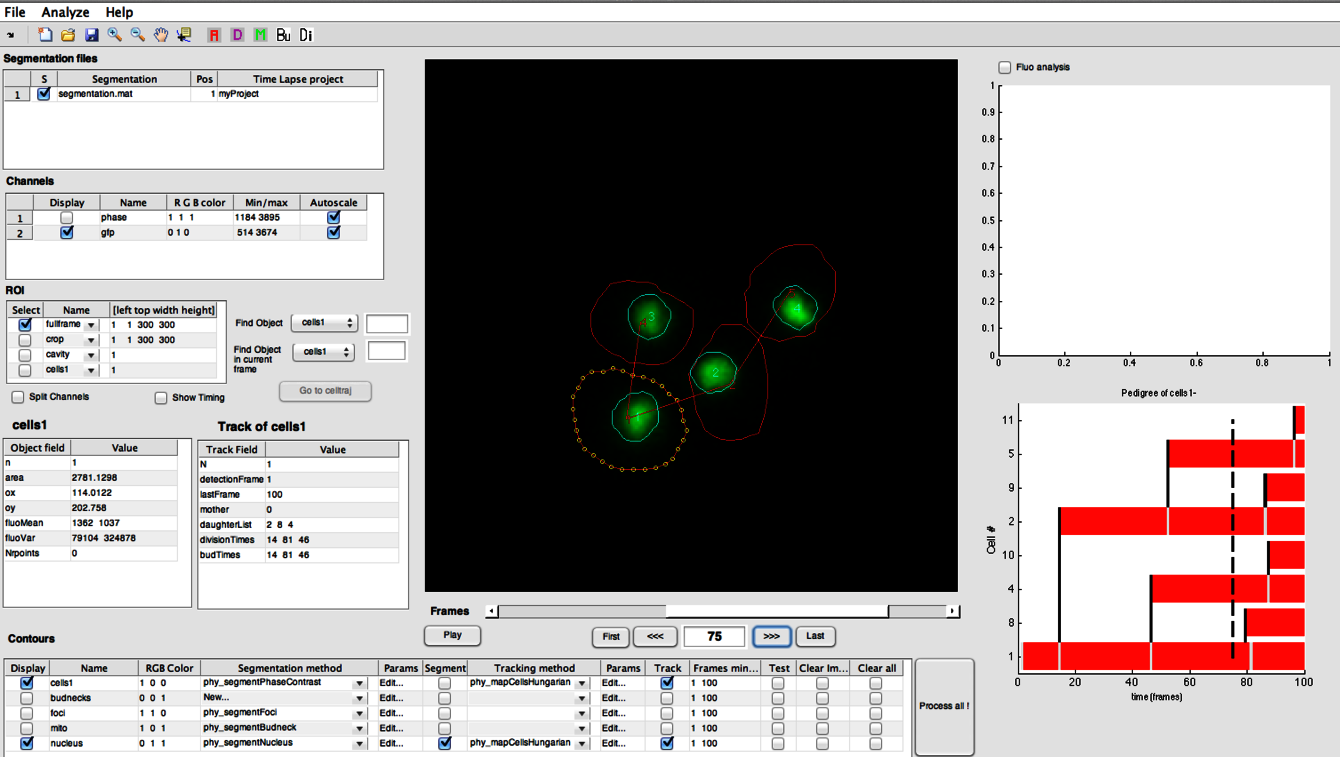
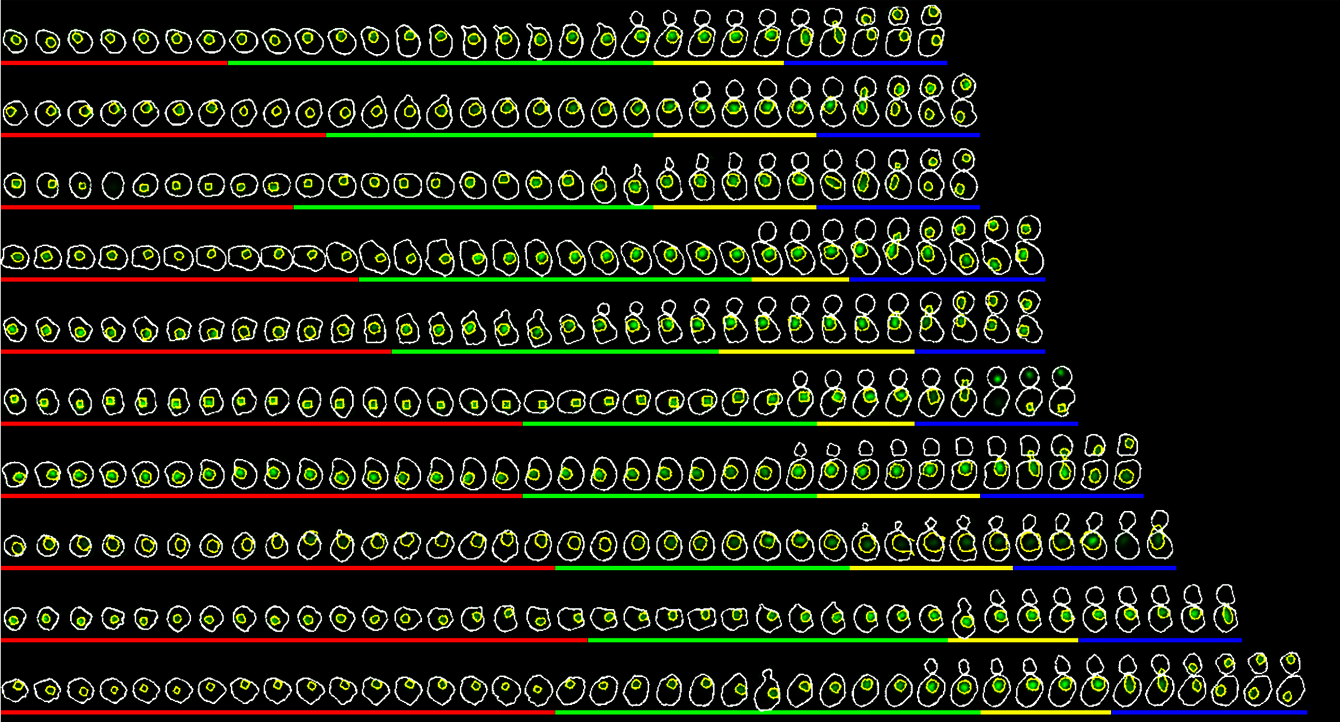
Matériel et techniques du laboratoire
L’équipe utilise les méthodes classiques de génétique moléculaire de la levure (transformation, croisements, etc.) pour générer des souches mutantes (dégron auxine dépendant, mutants conditionnels, expression conditionnelle) et marqueurs fluorescents (sondes ratiométriques redox ou pH, protéines de fusion, timer fluorescent, etc.).